SynBio Column Recent Research Roundup | RRR EP. 03
[SynBio RRR EP. 03]
ตลอดหลายปีที่ผ่านมา หลายคนคงเคยได้ยินเกี่ยวกับ “พืชดัดแปลงพันธุกรรม” (GMOs) ไม่มากก็น้อย (ถ้าไม่เคยก็ต้องตามอ่านงานของพวกเราบ่อย ๆ ละ) ตั้งแต่ความพยายามทำให้ผลผลิตทางการเกษตรมีเนื้อเยอะขึ้น ไปจนถึงต้นข้าวที่ทนร้อน ทนหนาว ทนเค็มได้ดีขึ้น แต่ไม่ว่าจะด้วยวิธีใด กระบวนการดัดแปลงพันธุกรรมที่ผ่านมา ส่วนใหญ่แล้วล้วนเป็นการ “คัดลอก” สิ่งที่เราต้องการจากสายพันธุ์ที่ใกล้เคียง หรือสิ่งมีชีวิตอื่น ๆ นำมาใส่ลงไปในสิ่งมีชีวิตเป้าหมาย เหมือนกับการยืมกิ่งไม้ชนิดอื่นมาปักชำลงในต้นที่เราต้องการ ตัวอย่างที่เกิดขึ้นจริง ๆ แล้วก็เช่นการสร้างต้นมะเขือเทศที่สามารถทนต่อแมลงได้ (1) นักพัฒนาพันธุ์พืชก็ได้ไปตามหายีนส์ของแบคทีเรียที่สามารถสร้างสารที่แมลงไม่ชอบมาได้ จากนั้นก็นำไปผ่านกรรมวิธีต่าง ๆ เพื่อให้ต้นไม้นี้สามารถเก็บเอายีนส์แบคทีเรียนี้ไว้ และสามารถสร้างสารที่แมลงไม่ชอบได้ในที่สุด
แต่แน่นอนว่าวิธีการที่กล่าวมาไม่ใช่เรื่องง่ายเลย ส่วนใหญ่ต้องลองผิดลองถูกหลายต่อหลายครั้ง การตัดต่อพันธุกรรมแต่ละครั้งก็ใช้เวลาค่อนข้างนาน ซึ่งบางทีเราก็พบข้อจำกัดมากมายในธรรมชาติ ไม่ว่าจะเป็นการตัดต่อที่ไม่สมบูรณ์ (เช่น ดัดแปลงไม่ครบทุกโครโมโซม ลองดูตัวอย่างที่ EP. 2 ได้ครับ) หรือแม้ว่าการตัดต่อจะเป็นไปด้วยดี แต่ผลลัพท์ที่ออกมาก็อาจจะไม่ได้มีประสิทธิภาพเท่ากับที่คาดหวังไว้ หรือแม้กระทั่งในบางกรณี การนำเอายีนใหม่ ๆ เข้าไปในสิ่งมีชีวิตนั้นก็อาจจะกระบวนการอื่น ๆ ที่จำเป็นต่อการดำรงชีวิต จนกระทั่งสิ่งมีชีวิตดัดแปลงนั้น ๆ ไม่สามารถเจริญเติบโตหรือทำงานดั้งเดิมได้ดี ปรากฎการณ์นี้เราเรียกกันว่า metabolic burden (2) ที่มากจนเกินทน
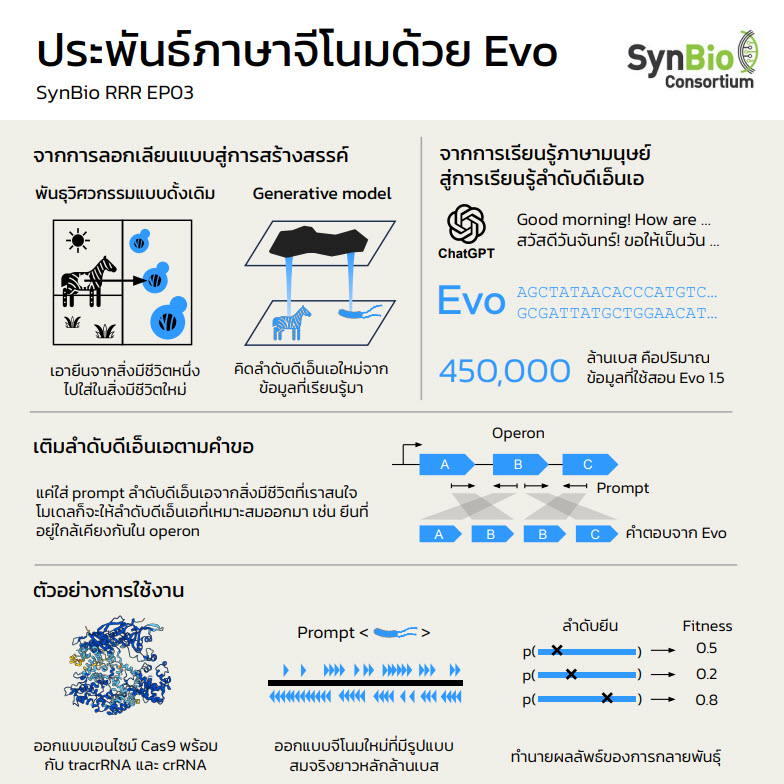
เมื่อปี 2018 นักวิทยาศาสตร์รางวัลโนเบล Frances Arnold ได้อธิบายว่า “ทุกวันนี้ ในทางปฏิบัติ เราสามารถอ่าน เขียน และแก้ไขลำดับดีเอ็นเอได้แทบทุกอย่าง แต่เรายังไม่สามารถ ‘ประพันธ์’ (Compose) มันได้” Frances Arnold ได้รับรางวัลโนเบลจากการนำเสนอไอเดียที่มีชื่อว่า “การกำกับวิวัฒนาการ (Directed Evolution)” (3) ซึ่งปัจจุบันเป็นกระบวนการที่ถูกใช้อย่างแพร่หลายในวงการชีววิทยาสังเคราะห์ในปัจจุบัน ไอเดียหลักของ Directed Evolution คือการสร้างต้นแบบที่มีการกลายพันธ์ูจากเป้าหมายเริ่มต้น แล้วคัดเลือกการกลายพันธุ์ที่ต้องการ คล้ายกับการบีบให้วิวัฒนาการนั้นเกิดขึ้นตามการกำกับของนักวิทยาศาสตร์อย่างพวกเรา ยกตัวอย่างเช่น หากเรามีข้อมูลว่าโปรตีนชนิดหนึ่งมีผลต่อการทนความร้อนของพืช เราก็สามารถสร้างโปรตีนจำนวนมากที่เกิดจากการตั้งใจทำให้โปรตีนนั้นกลายพันธ์ในหลาย ๆ รูปแบบ แล้วจึงเลือกตัวที่ทนความร้อนได้มากขึ้นมาเก็บไว้เพื่อทำซ้ำเป็นลำดับถัดไป การทดลองแนวนี้อาศัยการลองผิดลองถูกเยอะมาก ๆ โดยที่ในระดับโปรตีนแล้วอาศัยการเปลี่ยนแปลงเพียงรอบละไม่กี่ตำแหน่งเท่านั้น หากจะสร้างโปรตีนพันธุ์ใหม่ที่แตกต่างไปสัก 10 จุดอาจจะใช้เวลานับปีกว่าจะได้โปรตีนพันธุ์ใหม่นี้ออกมา บทความนี้จะยกกระบวนการใหม่ที่พยายามแก้ปัญหาตรงนี้ด้วยเทคโนโลยีเปลี่ยนโลกอย่าง Generative AI
โลกที่เปลี่ยนไปเมื่อปี 2022 (Advent of ChatGPT)
ไอเดียของ AI อาจจะอยู่คู่กับคนเรามาตั้งแต่ปี 1927 ที่ถูกพูดถึงในหนังเรื่อง Metropolis (นานมากขนาดที่แม่ของผู้เขียนก็ยังเกิดไม่ทันเลย แต่ไม่มีใครเก่งสู้แม่ผมได้แน่นอน :P) ซึ่งจากนั้นคนเราก็อยู่กับจินตนาการต่าง ๆ ไม่ว่าจะเป็น AI ที่อยู่ในหนัง Sci-fi หรือหนังสยองขวัญที่พูดถึงเรื่อง AI จะครองโลกก็ตาม จนเมื่อไม่นานมานี้มนุษยชาติก็เพิ่งที่จะเข้าใจพลังของสิ่งที่เรียกว่า Generative AI เมื่อตอนปี 2022 จากการที่บริษัท OpenAI ได้เปิดตัว ChatGPT หรือโมเดลภาษาขนาดใหญ่ที่สามารถโต้ตอบบทสนทนาได้ใกล้เคียงกับมนุษย์ และมีพัฒนาการอย่างต่อเนื่องผ่านมาหลากหลายเวอร์ชั่นทั้งในแง่ความฉลาดและความคิดสร้างสรรค์เรื่อยมา ไม่ว่าจะเป็นการแต่งนิยาย แต่งกลอน หรือแม้กระทั่งสูตรอาหาร (ถึงแม้จะไม่การันตีในระดับของความอร่อย แต่สูตรทั่วอินเตอร์เน็ตก็เป็นเช่นนั้นอยู่แล้ว เอาจริง ๆ protocol ในงานวิจัยวิทยาศาสตร์ก็ประมาณนี้แหละ!!!)
ก่อนจะกลับมาที่เรื่องราวของการประพันธ์จีโนม เราอยากจะยกเบื้องหลังความสำเร็จของ ChatGPT อย่างองค์ความรู้พื้นฐานที่ประกอบกันเป็นโมเดล AI ที่คุยกันอยู่ข้างหลังจอที่เราเห็นโดยเฉพาะกลุ่มที่เราเรียกว่า Transformer (4) (ไม่ใช่รถต่างดาว) ถ้าจะให้อธิบายอย่างง่ายก็คือเป็นโมเดลที่ถูกสร้างมาให้เหมาะกับการใช้งานกับข้อมูลที่เป็นลำดับ เช่น ข้อมูลทางภาษาที่มีลักษณะจากการอ่านจากทางซ้ายไปทางขวานั่นเอง (ขวาไปซ้ายก็ได้นะ แต่ต้องปรับจูนนิดหน่อย) แต่แน่นอนว่าภาษาคนไม่ใช่สิ่งเดียวที่มนุษย์อยากลอกเลียนแบบ จริง ๆ แล้วข้อมูลที่เป็นลำดับนั้นยังอยู่ในรูปแบบอื่น ๆ อีกมาก ไม่ว่าจะเป็นข้อมูลของลำดับกรดอะมิโนในโปรตีน หรือข้อมูลกลุ่มคลื่นสัญญาณอย่างคลื่นไฟฟ้าสมองและคลื่นไฟฟ้าหัวใจ และข้อมูลที่เราจะพูดถึงกันในวันนี้ก็คือข้อมูลทางพันธุกรรม หรือข้อมูลจีโนมอย่างลำดับของดีเอ็นเอนั่นเอง
จากการดัดแปลงและลอกเลียนแบบสู่การออกแบบและสร้างสรรค์
ถ้าหากเราพิจารณาที่ข้อมูลของจีโนม ชุดข้อมูลที่ใกล้เคียงที่สุดก็คงหนีไม่พ้นข้อมูลลำดับกรดอะมิโนของโปรตีนที่หากได้ติดตามวงการวิทยาศาสตร์อยู่เรื่อยก็น่าจะเคยได้ยินชื่อของงานวิจัยเปลี่ยนโลกอย่างโมเดล AlphaFold (5) ของทีม Google Deepmind (ทีมเดียวกันกับที่สร้าง Alpha Go มาล้มแชมป์โลกหมากล้อม) ที่ออกมาสู่สาธารณะเมื่อปี 2021 โมเดล AlphaFold นี้เรียนรู้การพับของลำดับโปรตีนให้เป็นโครงสร้างต่าง ๆ ที่สามารถนำเอามาประยุกต์ใช้ในเชิงการแพทย์หรือการหาโครงสร้างของเอนไซม์ต่าง ๆ เพื่อออกแบบยาใหม่ ๆ ที่สามารถเพิ่มประสิทธิภาพหรือยับยั้งได้นั่นเอง ก่อนที่ AlphaFold จะออกมาให้ใช้งาน ในอดีตการหาโครงสร้างของโปรตีนนั้นต้องพึ่งการตกผลึกโปรตีนซึ่งนอกจากจะใช้ต้นทุนสูงแล้ว ยังมีความไม่แน่นอนในการตกผลึกโปรตีนติดพันมาด้วย นักชีววิทยาโครงสร้างอาจจะใช้เวลาหลายปีกว่าจะได้โครงสร้างของโปรตีนชิ้นหนึ่งออกมา ในขณะที่ AlphaFold ช่วยทำนายได้ภายในพริบตาพร้อมกับความแม่นยำที่ใกล้ความจริงไปเสียทุกที งานวิจัยด้านการพับโปรตีนนี้ทำให้ทีมของ Google Deepmind (John Jumper และ Demis Hassabis) ได้รับรางวัลโนเบลในปีที่ผ่านมา (2024) ร่วมกับงานออกแบบโปรตีนด้วยคอมพิวเตอร์จากทีมของ David Baker จาก University of Washington
หลังจากการประสบความสำเร็จอย่างเหนือชั้นของ AI เพื่อทำนายและออกแบบโปรตีน หลายทีมวิจัยก็มีความพยายามที่ในการที่จะสร้างโมเดลเพื่อศึกษาข้อมูลของจีโนม ไม่ว่าจะเป็น Nucleotide Transformer โดยสตาร์ทอัพของทางอังกฤษ (6) หรือ GenSLMs ของทางทีมมหาวิทยาลัยชิคาโก (7) แต่ทั้งสองโมเดลที่ถูกพูดถึงครั้งแรกเมื่อปี 2023 ยังไม่ได้รับการถูกทดสอบในห้องปฏิบัติการมากนัก จนกระทั่งทีมจากมหาวิทยาลัยสแตนฟอร์ด นำโดย Brian Hie ผู้ซึ่งเคยอยู่ในทีมของ Meta AI ของ Facebook มาก่อน ได้ทำการเปิดตัวโมเดล Evo ที่ได้เรียนรู้ในจีโนมของกลุ่ม Prokaryote จำนวนมาก (8) ซึ่งสาเหตุของการเริ่มด้วย Prokaryote นั้นก็หนีไม่พ้นความสะดวกในการทดลองในห้องแล็บจริง เนื่องด้วยการกระบวนการวิศวกรรมง่ายอยู่ (ที่แปลว่าไม่ง่ายเท่าไร) ขนาดที่เล็กกว่า และกระบวนการทดสอบใช้เวลาน้อยกว่า ทำให้ทางทีมสามารถนำโมเดลที่สร้างขึ้นไปลองทดสอบได้จริงในเวลาไม่นานนัก
ในส่วนของโมเดลนั้น AI ได้เรียนรู้ผ่านข้อมูลลำดับเบสของ Prokaryote จำนวน 3 แสนล้านลำดับในเวอร์ชั่นแรก และ 4.5 แสนล้านลำดับในเวอร์ชั่น 1.5 ซึ่งเป็นการเรียนรู้ในรูปแบบเดียวกันกับโมเดลในเชิงภาษา (9) ซึ่งโดยส่วนมากเรียนรู้ด้วยการทำสิ่งที่เรียกว่าการทำนายคำถัดไป (Next Token Prediction) หรือการฝึกให้โมเดลเติมคำในช่องว่าง (Masked Language Modeling) แต่ครั้งนี้เปลี่ยนจากการเรียนภาษาเป็นคำๆเป็นการเรียนภาษาของ DNA เพียงเท่านั้นเอง ซึ่งก็ประกอบไปด้วยการเติม A T C G ให้ถูกต้อง สิ่งทางทีมวิจัยก็ได้ทำการประเมินผลลัพธ์ในคอมพิวเตอร์ในโจทย์ต่าง ๆ ดังนี้
สร้างพิษและยาต้านพิษ (Generation of Toxin-AntiToxin Systems)
โดยเรื่องหนึ่งที่ได้ถูกนำมาทดลองก่อนก็คือการให้โมเดลสังเคราะห์ลำดับเบสสำหรับการสร้างระบบพิษ-ยาต้านพิษ (Toxin-AntiToxin) ในแบคทีเรีย ระบบพิษต้านพิษที่ว่านี้ก็มีไว้สำหรับการป้องกันตัวจากไวรัสในแบคทีเรีย หรือ Bacteriophage (เรียกสั้น ๆ ว่า Phage) โดยหลักการทำงานระบบนี้คือ การแสดงออกของยีนสองชุดที่อยู่ติด ๆ กัน (ประเด็นนี้สำคัญมากในระบบการทดสอบ AIT ที่จะมาเติมคำในช่องว่าง หรือช่องข้าง ๆ) โดยยีนส์แรก (Toxin DNA) จะแสดงออกเพื่อการผลิตโปรตีนยาพิษ (Toxin protein) ส่วนอีกยีนหนึ่ง (AntiToxin DNA) จะสร้างออกมาเป็นโปรตีนยาต้านพิษ (AntiToxin protein) โดยคุณสมบัติพิเศษคือยาพิษเหล่านี้จะมีอายุขัยในเซลล์ที่ยืนยาวคงทน ในขณะที่ยาต้านพิษนั้นจะมีคงอยู่ไม่นาน หากว่าวันใดวันหนึ่งยีนส์ที่สร้างยาต้านพิษ (หรือทั้งสองยีน) หายไป หรือทำงานแย่ลอง แบคทีเรียกก็จะกลายเป็นตำนานที่ไร้ชีวิตไปโดยปริยาย เพราะเจ้าโปรตีนยาพิษมันไม่หายไปไหนง่าย ๆ บางกลุ่มของระบบพิษต้านพิษนี้ก็เชื่อว่าเกิดการทำงานขึ้นเมื่อ Phage นั้นทำให้การแปลรหัสพันธุกรรมอ่อนแอลง ก็จะส่งผลให้ตัวยาต้านพิษที่มีอายุขัยต่ำก็จะสลายไปและปล่อยให้พิษฆ่าแบคทีเรียตัวที่ถูก Phage โจมตีเพื่อป้องกันไม่ให้ Phage สามารถแพร่กระจายไปยังตัวอื่นได้นั่นเอง (อธิบายเพิ่มเติมเล็กน้อย Phage หรือไวรัสอย่าง SARS-CoV-2 นั้นจะเพิ่มปริมาณได้ก็ต่อเมื่อมันเข้าไปอยู่ในพาหะหรือเจ้าบ้าน และใช้ทรัพยากรของเจ้าบ้านในการเติบโตและเพิ่มปริมาณ ดังนั้นหากเจ้าบ้านชิงฆ่าตัวตายซะก่อน ก็จะไม่กลายเป็นตัวแพร่เชื้อให้เพื่อนในฝูงตายกันหมด)
ระบบพิษต้านพิษนี้ถูกนำมาปรับใช้ในวงการเทคโนโลยีชีวภาพและชีววิทยาสังเคราะห์ในรูปแบบของพลาสมิดโดยนักวิจัยการมีอยู่ของชุดพิษต้านพิษนี้ทำให้ประชากรของแบคทีเรียต้องพึ่งพาพลาสมิดในการมีชีวิตรอด เดิมทีแล้วเมื่อเราอยากให้แบคทีเรียผลิตสารบางอย่างจากยีนที่เราใส่เข้าไป นักวิจัยมักจะคัดเลือกแบคทีเรียที่มีพลาสมิดด้วยการใช้การดื้อยาปฏิชีวนะ (Antibiotic Resistance) ซึ่งในทางปฏิบัติแล้วมีต้นทุนที่สูงและมีความเป็นไปได้ที่แบคทีเรียรุ่นลูกจากการแบ่งตัวอาจจะทำพลาสมิดสูญหายระหว่างทาง (ไอ้พี่ชายมันเอาไปหมด) โดยเฉพาะถ้าเป็นการผลิตที่ไม่ได้ช่วยให้แบคทีเรียมีชีวิตที่ดีขึ้นด้วยละก็ แบคทีเรียที่ไม่มีพลาสมิดก็มักจะโตเร็วกว่าตัวที่ต้องแบกยีนชุดนั้นเอาไว้ด้วยและท้ายที่สุดก็จะก้าวขึ้นมาเป็นประชากรส่วนใหญ่ในที่สุดแล้วปรับเปลี่ยนโครงสร้างประชากรในที่สุด การมีระบบพิษต้านพิษทำให้แบคทีเรียที่สูญเสียพลาสมิดตายลงไปในที่สุด เหลือแต่พวกที่ทำงานหนักเผื่อผลิตพิษมาต้านพิษอย่างสม่ำเสมอเท่านั้นจึงจะอยู่รอด
ไอเดียของทางทีมวิจัยจาก Arc Institute ในการให้โมเดลสร้างจีโนมใหม่ก็คล้าย ๆ กับ ChatGPT นั่นแหละ แต่แทนที่จะตั้งคำถามด้วยภาษาเขียนแบบทีเราแชทคุยกับบอท ก็เปลี่ยนเป็นการให้ข้อมูลของยาต้านพิษไปเหมือนเป็นคำถาม แล้วให้โมเดลเติมข้อมูลของพิษตัวใหม่ขึ้นมาได้ (เนื่องจากสองยีนนี้อยู่ใกล้กันในธรรมชาติ การให้ข้อมูลส่วนหนึ่งจะสามารถชี้นำไปสู่การสร้างคู่ของมันขึ้นมาได้) และเช่นกัน ถ้าให้ข้อมูลของยาพิษไปเป็นคำถาม โมเดลก็น่าจะสามารถสร้างยาต้านพิษตัวใหม่ที่เหมาะสมสำหรับยาพิษตัวนั้น ๆ ได้ ความน่าสนใจของโมเดลนี้คือการที่มันสามารถผลิตทั้งพิษและยาต้านพิษตัวใหม่ขึ้นมาได้ โดยทางทีมเริ่มจากการสังเคราะห์พิษตัวใหม่ขึ้นมาก่อนจากการให้ยาต้านพิษที่มีในธรรมชาติ ซึ่งจากการทดสอบในห้องแลปพบว่าพิษตัวใหม่นี้สามารถฆ่าแบคทีเรียได้ราว ๆ 80% และมีโครงสร้างที่แตกต่างจากยาพิษคู่หูตัวเดิมของยาต้านพิษที่ป้อนเข้าไป หลังจากนั้นเมื่อนำยาพิษชนิดใหม่นี้มาป้อนเป็นคำถาม โมเดล Evo นี้ก็สามารถออกแบบยาต้านพิษชนิดใหม่ที่มีความจำเพาะกับยาพิษตัวใหม่นี้ได้เช่นกัน ท้ายที่สุดก็สามารถสร้างคู่พิษต้านพิษระบบใหม่ที่ไม่เคยมีมาก่อนในธรรมชาติออกมาได้!!! (อย่างกับพิษประจิม นับถือ นับถือ)
สร้าง CRISPR-Cas และ Anti-CRISPRs
หลังจากที่เรียนตำราพิษต้านพิษสำเร็จแล้ว เคล็ดวิชาถัดไปที่จะให้เจ้า Evo ได้ฝึกฝนดูก็หนีไม่พ้นการจำลองการสร้างระบบซึ่งจำเป็นต้องใช้ทั้งรหัสของ RNA และโปรตีนในการทำงานอย่าง CRISPR-Cas ที่มาจากระบบภูมิคุ้มกันของแบคทีเรียในการป้องกันตัวจาก Phage (ไวรัสของแบคทีเรีย) โดยไวรัสเหล่านี้มักจะแพร่กระจายจากการแทรกชิ้นส่วน DNA ของตัวเองให้เข้าไปยังแบคทีเรีย แล้วทำให้แบคทีเรียอ่าน DNA ดังกล่าวแล้วผลิตชิ้นส่วนของไวรัสขึ้นมา กระบวนการที่ระบบ CRISPR-Cas ใช้ก็คือจดจำชิ้นส่วน DNA ของไวรัสแล้วจ้องที่จะตัดชิ้นส่วน DNA แปลกปลอมเหล่านั้นของไวรัสที่เข้ามาจู่โจม ความพิเศษของมันอยู่ที่มันอาศัยการจับ DNA แปลกปลอมผ่านการสร้าง RNA นักวิทยาศาสตร์หลากหลายทีมจึงได้นำเอาชิ้นส่วน RNA เหล่านี้มาดัดแปลงพร้อมกับโปรตีน Cas ทั้งหลายมาใช้ประยุกต์ในการตัดต่อยีนที่สนใจนั่นเอง ยกตัวอย่างง่าย ๆ ก็คือเอาไปใส่ในเซลล์มนุษย์ให้มันตัดต่อยีนของคนซะเลย แม้ว่าปกติมันจะทำงานได้ดีในเซลล์ของแบคทีเรียแต่นักวิจัยทั่วโลกก็ได้ลงมือดัดแปลงให้มันใช้งานในเซลล์มนุษย์จนได้ นำไปสู่รางวัลโนเบลในปี 2020 ให้กับ Emmanuelle Charpentier และ Jennifer Doudna หากอยากอ่านเรื่องนี้เพิ่มลองไปดูใน RRR EP02 ได้เลยครับ สำหรับในบทความนี้ ความน่าสนใจอยู่ที่ Guide RNA (gRNA) ที่ใช้บ่งชี้ DNA ที่ตัวโปรตีน Cas ต้องไปตัดที่มีความหลากหลายอยู่มาก ในระบบ CRISPR-Cas ที่มีอยู่มากกว่า 6 กลุ่มใหญ่เข้าไปแล้ว (10) ก็มีโครงสร้างของ gRNA แต่มีลักษณะแตกต่างกัน นอกจากนี้ยังมีระบบคล้าย ๆ กันอย่าง Argonaut หรือ TIGR-Tas ที่เพิ่งจะออกมาเปรียบเทียบกันอีก ในประเด็นของการตามหา CRISPR-Cas ใหม่ ๆ นั้นจึงมีความสำคัญมากในการสร้างระบบการตัดต่อยีนที่จำเพาะ (และโดยเฉพาะในเชิงพาณิชย์) ยกตัวอย่างเช่นหากเราต้องการจะนำ CRISPR-Cas ไปใช้ในการรักษาโรคอย่างการให้มันตัดต่อยีนจำเพาะในร่างกายของมนุษย์ เราก็คงอยากให้มันมีการตัดที่มีความแม่นยำสูงมาก ๆ และตัดเฉพาะส่วนที่ต้องการให้มีการตัดต่อเท่านั้น ไม่ไปตัดที่อื่นมั่วซั่วจนเซลล์มนุษย์กลายพันธุ์เป็นเอเลี่ยน ในจุดนี้แม้ว่าจะมีการประยุกต์ใช้เพื่อรักษาโรคทางพันธุกรรมอยู่ในหลายกรณี (11) แต่การตัดต่อเซลล์ต้นกำเนิดหรือ Germline ก็ยังเป็นที่ถกเถียงกันเป็นอย่างมาก การตัดต่อ embryo ของคนครั้งแรกนั้นได้รับวิจารณ์ในแง่ลบเป็นอย่างมากว่ามันเกิดขึ้นเร็วเกินไปกว่าที่เทคโนโลยีและวงการจะพร้อมรับมือกับผลที่ตามมา (12) — เริ่มออกนอกเรื่อง แค่นี้พอละกัน
กลับมาที่การนำโมเดล Evo มาใช้สร้างระบบ CRISPR-Cas ทางทีมได้ทำการทำสิ่งที่เรียกว่าการปรับแต่งโมเดล (Finetuning) โดยการนำข้อมูลจาก CRISPR-Cas ชนิดต่าง ๆ มาให้โมเดลได้ทำการเรียนรู้โดยเฉพาะเจาะจงมากขึ้น โดยการอาศัยให้โมเดลเรียนรู้จากข้อมูล CRISPR-Cas ในรูปแบบต่าง ๆ เพื่อให้รู้มากขึ้นว่ายีน CRISPR-Cas นั้นควรจะมีหน้าตาอย่างไร และควรเขียนออกมาแบบไหน แล้วจากนั้นนำโมเดลที่มีการปรับแต่งนี้ไปใช้ในการสร้าง CRISPR-Cas ตัวใหม่ โดยทำการสร้างขึ้นมากว่า 2 ล้านรูปแบบ แต่เนื่องจากจะมีการนำไปทำผลลัพท์ต่อ จึงต้องมีเกณฑ์การเลือกตัวที่น่าจะสามารถไปสร้างได้จริงและมีความแตกต่างจากตัวที่พบในธรรมชาติพอสมควร และอีกหนึ่งเกณฑ์ที่ถูกนำมาใช้ในการคัดเลือกก็คือคะแนนที่มาจากความมั่นใจในโครงสร้างของ Alphafold3 (pLDDT) นั่นเอง โดยจากทั้งหมดแล้วมี CRISPR-Cas ที่ถูกนำไปทดลองในห้องแลปจริง ๆ อยู่ทั้งหมด 11 ตัว และหนึ่งในนั้นก็มีประสิทธิภาพเทียบเท่ากับตัว CRISPR-Cas9 ที่พบในธรรมชาติได้เลย แม้ว่าหลายคนอาจจะมองว่าเป็นตัวเลขที่ไม่เยอะเท่าไหร่นัก แต่ในแง่การศึกษาในจากสร้างยีนชนิดใหม่แล้วนั้น การที่เลือกตัวสังเคราะห์มา 11 ตัวแล้วมีตัวที่สำเร็จ 1 ตัวนั้นก็นับเป็นอัตราความสำเร็จที่ค่อนข้างสูงเลยทีเดียว
อย่างไรก็ตาม CRISPR-Cas ที่ถูกสร้างขึ้นมาใหม่ด้วย Evo นี้มีลักษณะคล้ายกับ CRISPR-Cas ที่ถูกนำไปปรับแต่งโมเดลอยู่ที่ราว 80% ซึ่งถือว่าค่อนข้างสูงอยู่สำหรับการสร้างยีนใหม่ (เอ๊ะ มันใหม่จริงปะนะ) ซึ่งในปัจจุบันการทดลองการสร้างโปรตีนใหม่นั้นจะมีส่วนที่ยากคือการค้นหายีนหรือโปรตีนตัวใหม่ ๆ ที่มีความเหมือนกับข้อมูลที่พบในธรรมชาติไม่ถึง 30% ดังนั้น หนึ่งในเป้าหมายของการทดลองที่ถูกเลือกมาเพื่อทดโมเดล Evo จึงมุ่งไปที่การสร้างยีนที่แตกต่างจากในธรรมชาติในระดับนี้ ทีมวิจัยจาก Arc Institute เลือกสร้างยีนกลุ่ม Anti-CRISPRs (Acr) ซึ่งเป็นยีนในไวรัสที่มีหน้าที่ในการต่อต้านระบบ CRISPR-Cas ของแบคทีเรียเพื่อให้มันสามารถบุกโจมตีแบคทีเรียเหล่านี้ได้เช่นเดิม โปรตีนในกลุ่ม Acr นี้มีความหลากหลายที่ค่อนข้างสูง และมีจำนวนที่ถูกค้นพบแล้วก็ไม่เยอะเทียบกับ CRISPR-Cas สาเหตุหนึ่งก็เพราะว่ามันค้นพบยากกว่า (ยีนมันอยู่ในไวรัส) ทางทีม Arc Institute จึงได้ลองนำข้อมูลบางส่วนของยีน Acr ไปให้โมเดล Evo สร้างต่อจำนวนทั้งหมด 3180 ลำดับ แล้วผ่านการคัดกรองด้วยวิธีการทางคอมพิวเตอร์จนเหลือ 84 ตัว เมื่อทำไปทดสอบในห้องแลปพบว่ามีทั้งหมด 14 ตัวที่มีความสามารถในการยับยั้ง CRISPR-Cas9 ได้ และที่น่าสนใจไปกว่านั้นคือมี 3 ตัวในนี้ที่มีความเหมือนกับโปรตีนในธรรมชาติไม่ถึง 30% นอกจากนี้ 2 ตัวในกลุ่มนี้ยังไม่พบโปรตีที่คล้ายคลึงในธรรมชาติได้เลย (นี่มันชีววิทยาสังเคราะห์ชัด ๆ) ส่วนอีกหนึ่งตัวพบความคล้ายมากที่สุดก็มีลำดับโปรตีนคล้ายกับตัวในธรรมชาติไม่เกิน 25% เท่านั้น นับว่าเป็นโปรตีนที่ใหม่หลุดโลกมาก ๆ ระดับจักรวาลเลยทีเดียว
แล้วโมเดลในสิ่งมีชีวิตอื่น ๆ ละ มีอะไรน่าสนใจตามมาอีกมั้ย
จริง ๆ แล้วในวงการนั้นมีความพยายามจากหลากกลุ่มในการสร้างโมเดลสำหรับการเรียนรู้ข้อมูลจีโนมของสิ่งมีชีวิตอื่น ๆ โดยเฉพาะมนุษย์ เพื่อให้เราเข้าใจโรคต่าง ๆ มากขึ้น ไม่ว่าจะเป็นโรคหายากทางกรรมพันธุ์ หรือโรคมะเร็ง (ที่แท้จริงแล้วก็เกิดจากเซลล์ในร่างกายของเราเกิดการกลายพันธุ์ที่ทำให้หยุดแบ่งตัวไม่ได้จนเกิดเป็นก้อนเนื้อ) ซึ่งสาเหตุที่จีโนมของมนุษย์นั้นมีความยากมากกว่าจีโนมของแบคทีเรียก็หนีไม่พ้น 3 ประเด็นหลัก ๆ (13) ได้แก่
1. มันใหญ่มาก: มนุษย์นั้นมีจำนวนยีนมากกว่าแบคทีเรียอยู่ที่ประมาณ 25 เท่า แต่ที่ยิ่งไปกว่านั้น เรามีจำนวนเบสของ DNA มากกว่าแบคทีเรียอยู่มากกว่า 650 เท่าเลยทีเดียว
2. มันซับซ้อน: มนุษย์มีระบบของการควบคุมการแสดงออกของยีนที่ซับซ้อนกว่าแบคทีเรียมาก ๆ ในระบบของแบคทีเรียอย่าง E. coli นั้น จีโนมกว่า 90% ถูกนำไปแปลเป็นโปรตีน และอีก 10% นั้นมีหน้าที่ในการควบคุมการแสดงออกของยีนอีกที แต่ในมนุษย์นั้นจีโนมที่ถูกนำไปแปลเป็นโปรตีนนั้นมีแค่ประมาณ 3% เท่านั้น กว่า 97% นั้นมีหน้าที่ในการควบคุมการแสดงออก
3. มันซ้ำ ๆ: มนุษย์นั้นมีจีโนมที่มีรูปแบบซ้ำซ้อนกันสูงมาก โดยที่บางส่วนของ DNA ของคนเรานั้นสามารถซ้ำกันได้สูงสุดราวหลักแสนครั้งเลยที่เดียว
ซึ่งหลังจากทีมงานได้นำงาน Evo 1 และ Evo 1.5 มาพูดคุยกันใน Journal Club ได้เพียงสามวัน… ทีม Arc Institute ก็ออกโมเดล Evo 2.0 ซึ่งเรียนรู้จากสิ่งมีชีวิตทุกประเภทออกมา (14) (แล้วทำให้ทีมงานคิดไม่ตกว่าจะเขียนอธิบายต่อยังไงดี)
แต่ในการปล่อยงานนี้ยังอยู่ในระดับการทำโจทย์ที่เป็นการทำเชิงคอมพิวเตอร์อยู่ และเปิดโอกาสให้ทีมอื่น ๆ นำเอาโมเดลไปประยุกต์ใช้ในการออกแบบการทดลองได้ทันที ยกตัวอย่างเช่นการดูว่า Evo 2.0 นั้นสามารถบอกการกลายพันธุ์ที่น่าจะก่อให้เกิดโรคในคนได้หรือไม่ คำถามที่ว่าจะเป็นในด้านการทดสอบว่ายีนที่สร้างจาก Evo 2.0 นั้นจะเป็นยังไง และยีนใหม่ ๆ นั้นจะมีประสิทธิภาพและการนำไปใช้งานแบบใดได้บ้างคงต้องรองานในอนาคตมาอธิลายเป็นลำดับถัดไป
สิ่งหนึ่งที่น่าสนใจคือการที่ทางทีมมี Arc Institute ตั้งใจตัดสิ่งมีชีวิตกลุ่มหนึ่งออกไปจากการเรียนรู้ของโมเดล Evo 2.0 นั้นคือสิ่งมีชีวิตกลุ่มไวรัสที่มีการแพร่พันธุ์ในสิ่งมีชีวิตนอกเหนือจากแบคทีเรีย เช่นไวรัสในมนุษย์หรือสัตว์ ประเด็นนี้เป็นประเด็นสำคัญของเรื่องความปลอดภัยทางชีวภาพ (biosecurity) และทางทีมก็ได้ทดลองด้วยการให้โมเดลลองเติมข้อมูลของไวรัสและพบว่าความสามารถของโมเดลนั้นไม่ต่างจากการเดาสุ่ม ทั้งนี้ก็เพื่อให้มั่นใจว่าโมเดลจะไม่มีการถูกนำไปใช้ในการสร้างไวรัสตัวใหม่ ๆ ที่มีโอกาสจะติดมนุษย์ได้นั้นเอง ส่วนตัวทีมงานชีววิทยาเหนือธรรมชาติก็ได้ไปพบกับ Brian Hie ที่เป็นนักวิจัยหลักของการสร้างโมเดล Evo จาก Arc Institute ในการประชุม Spirit of Asilomar (15) ที่เน้นการเสวนาเกี่ยวกับเทคโนโลยีชีวภาพในโลกอนาคต การจัดการความปลอดภัยของกระบวนการและข้อมูลต่าง ๆ ไปจนถึงผลกระทบที่อาจจะเกิดขึ้นจากการงานวิจัยแนวหน้าอย่าง Generative Genomic Model เหล่านี้ โดยมี AI in Biotechnology เป็นหนึ่งในห้าประเด็นหลักของการเสวนาครั้งนี้เลยทีเดียว การเผยแพร่ของ Evo เพียงไม่กี่สัปดาห์ก่อนการประชุมก็ทำให้เกิดแรงกระเพื่อมในการสนทนาไม่แพ้หัวข้ออื่น ๆ อย่างเซลล์เงาสะท้อนสังเคราะห์ (synthetic mirrored cell) เทคโนโลยีชีวภาพแบบเหนือการควบคุม (beyond biocontainment biotechnology) เชื้อก่อโรคและอาวุธชีวภาพ (pathongens and bioweapons) และการตีกรอบเทคโนลียีชีวภาพโลกอนาคต (framing future biotechnology) ซึ่งเป็นที่แน่นอนว่าวงการเทคโนโลยีชีวภาพและชีววิทยาสังเคราะห์คงต้องมีการเปลี่ยนแปลงอย่างก้าวกระโดดในอีกไม่กี่ปีข้างหน้าด้วยการสร้างข้อมูลใหม่ในสเกลนับล้านล้านเบสด้วยโมเดลอย่าง Evo แน่นอน
References
(13) Cooper, G. M. The Cell: A Molecular Approach. 2nd Edition; Sinauer Associates 2000, 2000.