Synthetic Biology: เมื่อมนุษย์กลายเป็นนักประดิษฐ์ชีวิต [EP.3]
*ภาพปกเป็นภาพเปรียบเทียบ E. coli เซลล์จุลินทรีย์ป่าที่ยังยุ่งเหยิง ไม่ได้รับการจัดการให้สวยงาม Syn3A แบคทีเรียมินิมัลที่มีแค่ฟังก์ชั่นเพื่อการดำรงชีวิต และ Sc2.0 ยีสต์เวอร์ชั่นใหม่ที่มีจีโนมถูกจัดเรียงอย่างเป็นระบบระเบียบโดยฝีมือมนุษย์ (ภาพ AI สร้างโดย ผศ. ดร.ป๋วย อุ่นใจ)
20 มกราคม 2025 คือวันที่ประวัติศาสตร์หน้าใหม่ถูกจารึกเอาไว้ นี่คือปัจฉิมบทของการออกแบบจีโนมใหม่ของยีสต์ และคือจุดเริ่มต้นของยุคแห่งการเขียนจีโนมของสิ่งมีชีวิตขึ้นมาเองโดยมนุษย์
เปเปอร์ Construction and iterative redesign of synXVI a 903 kb synthetic Saccharomyces cerevisiae chromosome จากทีมพันธมิตรนำโดยฮิวจ์ กูลด์ (Hugh Goold) เอียน พอลเซน (Ian Paulsen) และไอแซค พรีโทเรียส (Isak Pretorius) จากมหาวิทยาลัยแมคแควรี (Macquarie University) ที่เผยแพร่ออกมาในวารสาร Nature Communications ในวันนั้นตีพิมพ์รายงานความสำเร็จของการออกแบบและปรับแก้โครโมโซม XVI ซึ่งเป็นโครโมโซมสังคราะห์เส้นสุดท้ายในโครงการออกแบบจีโนมใหม่ให้ยีสต์หรือ Sc2.0 และนี่คือครั้งแรกที่มนุษยชาติสามารถเขียนจีโนมของสิ่งมีชีวิตยูคาริโอตขึ้นมาใหม่ได้ทั้งหมดแบบจริง ๆ จัง ๆ
และนี่ไม่ใช่สิ่งมีชีวิตที่มีจีโนมแบบมินิมัล (minimal genome) อย่าง Synthia ของจอห์น กลาส (John Glass) เครก เวนเทอร์ (Craig Ventor) และทีมจาก JCVI (J Craig Ventor Institute) ที่ตอนนี้ พัฒนาไปถึงเวอร์ชั่น 3A (synthetic Mycoplasma mycoides JCVI-syn3A) จนเหลือจีโนมเพียงแค่ 5 แสน 5 หมื่นเบส และยีนแค่ราว ๆ 500 ยีน (ซึ่งเล็กกว่าจีโนมของแบคทีเรีย Escherichia coli ที่เราใช้กันอยู่ทั่วไปในแลบถึงเกือบ 10 เท่า ) แต่เป็นจีโนมของยีสต์ เซลล์ยูคาริโอตที่มีความซับซ้อนอย่างที่สุด ขนาดจีโนมใหญ่โตมโหฬารถึง 12 ล้านคู่เบส ราว ๆ 6000 ยีน แบ่งกระจายออกไปเป็น 16 โครโมโซม แม้ว่าจุดมุ่งหมายจะคล้ายกันคือให้เข้าใจหลักการดีไซน์จีโนม (design principle) อย่างถ่องแท้ แต่ในรายละเอียด Syn3A และ Sc2.0 นั้นต่างกันแบบหน้ามือเป็นหลังมือ
Syn3A ผ้าใบผืนว่างกับการสรรสร้างค์สิ่งมีชีวิตฝีมือมนุษย์
“อะไรที่ผมสร้างขึ้นมาไม่ได้ ผมยังไม่เข้าใจ“ ริชาร์ด ไฟน์แมน (Richard Feynman) นักฟิสิกส์นามกระเดื่องเคยกล่าวไว้ และถ้าอยากเข้าใจชีวิต เราจะต้องออกแบบชีวิตได้ ในกรณีของ Syn3A หากอยากเข้าใจจริง ๆ ต้องสามารถออกแบบจีโนมทั้งหมดของมันขึ้นมาใหม่ได้จากศูนย์ แต่ถ้าทำไม่ได้ อย่างน้อยก็ต้องรู้ให้ได้ว่ามียีนอะไรบ้างที่สำคัญต่อชีวิต Syn3A จึงถูกออกแบบมาให้เป็นจุลินทรีย์ที่มินิมัลที่สุด คือมีสารพันธุกรรมเท่าที่จำเป็นแค่สามารถแบ่งเซลล์อยู่รอดได้และไม่เพี้ยนแค่นั้น
พวกเขาเปรียบ Syn3A เป็นเหมือนผ้าใบผืนว่าง ที่พร้อมจะรับทุกการปรับแต่ง ปฏิกิริยาในกระบวนการเมทาโบลิซึมก็มีแค่เพียงเท่าที่จำเป็นต่อการดำรงชีพและสืบต่อเผ่าพันธุ์ เอนไซม์ทุกตัวที่สร้างได้ และปฏิกิริยาส่วนใหญ่ก็ถูกศึกษามาแล้วอย่างทะลุปรุโปร่งแทบทั้งหมด ทั้งใน ด้านจลนพลศาสตร์ กลไกทางชีวเคมี การควบคุม หรือแม้แต่อัตราการเร่งปฏิกิริยา และด้วยความร่วมมือกับทีมวิจัยของอลิซาเบธ วิลลา (Elizabeth Villa) นักชีววิทยาเชิงโครงสร้างจากมหาวิทยาลัยแคลิฟอร์เนีย ซานดิเอโก (University of California San Diego) พวกเขาใช้เทคนิคการประกอบภาพความละเอียดสูงจากกล้องจุลทรรศน์อิเล็กตรอนที่เรียกว่า cryogenic electron tomography (cryoET) และสามารถทำแอตลาส (atlas) แสดงตำแหน่งของดีเอ็นเอทุกเส้นและเอนไซม์หลัก ๆ เกือบทุกตัวของเซลล์ Synthia ขึ้นมาใหม่ได้อย่างละเอียดเป็นสามมิติ
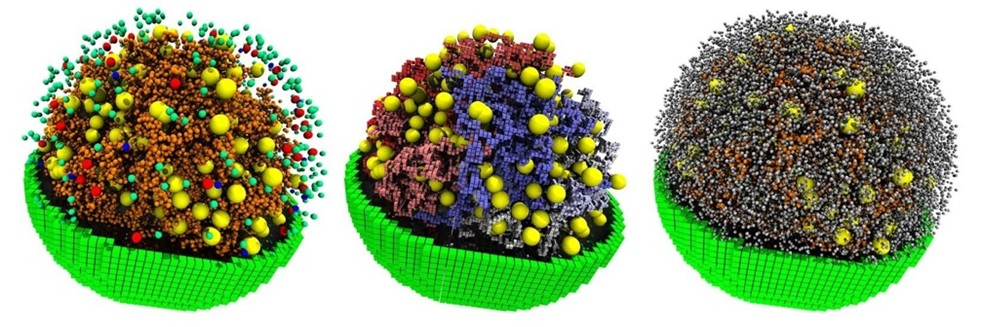
เมื่อรู้โครงสร้างของเซลล์ รู้ตำแหน่งของเอนไซม์ อีกทั้งยังรู้คุณสมบัติและกลไกในการทำงานของเอนไซม์ทั้งหมด พวกเขาก็สามารถสร้างแบบจำลองลูกบอลที่มีหน้าตาละม้ายคลายคลึงกับโมเดลจากเกม mine craft หรือที่หลายคนมักเรียกว่าแบบจำลองของเล่น (toy model) ที่ใช้ลูกบอลขนาดต่าง ๆ แทนเอนไซม์แต่ละชนิดขึ้นมา และกำหนดให้บอลแต่ละลูกมีคุณสมบัติเฉพาะตัวของแต่ละเอนไซม์เพื่อจำลองแบบพฤติกรรมเอนไซม์ภายในเซลล์ทั้งหมดในคอมพิวเตอร์ ที่น่าอัศจรรย์คือแบบจำลองของเล่นของพวกเขาช่วยให้นักวิทย์เห็นภาพกลไกการควบคุมการเกิดปฏิกิริยาชีวเคมีต่าง ๆ ในเมตาโบลิซึมภายในเซลล์ที่มีความมินิมัลอย่างที่สุด อีกทั้งยังสามารถทำนายการเปลี่ยนแปลงพลวัตของการแบ่งเซลล์และพฤติกรรมของเซลล์ที่ถูกปรับแต่งหรือกลายพันธุ์ไปได้อีกด้วย
ภาพแบบจำลองของเล่นจากห้องทดลองของไซดา ลูเธย์ ชูลเทน (Zaida Luthey-Schulten) จากมหาวิทยาลัยอิลลินอยด์ เออร์บานา แชมเปญ (Courtesy of University of Illinois Urbana Champaign)
นี่คืออนาคตแห่งเทคโนโลยีชีวภาพที่จะเปลี่ยนโฉมหน้าของเทคโนโลยีไปแบบพุ่งทะยานจนยากจะทำนายได้ เพราะถ้าเปรียบเซลล์มินิมัลนี้เป็นผ้าใบผืนว่างที่พร้อมรอจิตรกรมาแต่งเติมเพื่อสร้างนวัตกรรมพลิกโลก และเราสามารถจำลองแบบและทำนายได้ได้อย่างแม่นยำก่อนว่าผลงานชิ้นเอกที่จะสร้างสรรค์ขึ้นมานั้นจะมีลักษณะเป็นเช่นไร จะมีคุณสมบัติเป็นไปดังที่คาดไว้หรือไม่ในคอมพิวเตอร์ โดยไม่ต้องสร้างพวกมันขึ้นมาจริง ๆ ได้ ความก้าวหน้าของเทคโนโลยีออกแบบชีวิตก็จะพัฒนาไปได้รวดเร็วขึ้นอย่างมหาศาล และถ้าเอาเอไอมาบูรณาการด้วยแล้ว อนาคตแห่งเทคโนโลยีชีวภาพคงเป็นอะไรที่เหนือจินตนาการ
“ยีสต์” จากปริศนาแห่งอดีตถึงเทคโนโลยีปัจจุบัน
สำหรับโครงการยีสต์ Sc2.0 หรือยีสต์เวอร์ชั่น 2.0 นั้นกลับมีจุดมุ่งหมายที่แตกต่างไป จาก Syn3A อยู่มากโข มนุษย์รู้จักใช้ยีสต์มานับหมื่นปีตั้งแต่ก่อนที่จะรู้จักว่ายีสต์คืออะไรเสียอีก ทั้งการหมักเบียร์ kas ในอาณาจักรเมโสโปเตเมีย การหมักไวน์ข้าวในจีน ไปจนถึงการหมักขนมปังฟู (leavened bread) ในอียิปต์ ในยุคนั้นกลไกแห่งกระบวนการหมักยังคงเป็นที่ถกเถียงว่าแท้จริงเป็นแค่การเน่าเปื่อยแปรสภาพทางเคมี หรือมีจุลินทรีย์มาเกี่ยวข้อง การมีอยู่ของยีสต์เริ่มชัดเจนขึ้นเมื่อ แอนโทนี แวน ลิวเวนฮุค (Antonie van Leeuwenhoek) ส่องเห็นเซลล์ ยีสต์ภายใต้กล้องจุลทรรศน์ และเมื่อศึกษาโครงสร้างลักษณะของยีสต์อย่างละเอียด ในเวลาต่อมา ทีโอดอร์ ชวานน์ (Theodor Schwann) ก็สังเกตเห็นการแตกหน่อของเซลล์ยีสต์ เขาตีความว่ายีสต์นั้นเป็นสิ่งมีชีวิตและน่าจะเป็นจุลินทรีย์จำพวกเชื้อรา
แต่ทุกอย่างยังคงไม่มีข้อยุติ จวบจนกระทั่ง หลุยส์ ปาสเตอร์ (Louis Pasteur) พิสูจน์ว่ายีสต์คือพระเอกตัวจริงเบื้องหลังกระบวนการหมักแอลกอฮอล์ในไวน์ เบียร์และอาหารหมักอื่น ๆ มนุษย์ถึงได้รู้ว่ายีสต์ คืออะไร แต่นั่นก็หลังจากที่ได้ใช้ยีสต์และผลิตภัณฑ์ของยีสต์มายาวนานนับสหัสวรรษ และในปี 1883 อีมิล คริสเตียน แฮนเซน (Emil Christian Hansen) จากห้องทดลองคาร์ลส์เบิร์ก (Carlsberg laboratory) ก็สามารถแยกเชื้อยีสต์ออกมาให้บริสุทธิ์ได้สำเร็จซึ่งช่วยผลักดันความก้าวหน้าของวงการอาหารหมัก (โดยเฉพาะอย่างยิ่งเบียร์) ไปอย่างรวดเร็ว และเนื่องจากมีการกินการใช้มายาวนาน ยีสต์จึงได้รับการตีตราว่า GRAS (generally recognized as safe) หรือก็คือ “ปลอดภัย” ในการนำมาใช้อุปโภคและบริโภค และด้วยเหตุนี้ ยีสต์จึงถูกใช้อย่างแพร่หลายในหลายอุตสาหกรรม โดยเฉพาะอย่างยิ่ง ในวงการอาหาร และเชื้อเพลิง ส่วนในวงการวิจัย ยีสต์ก็เป็นหนึ่งในจุลินทรีย์ที่ได้รับความสนใจมากที่สุด และมีการศึกษามากที่สุดในเกือบทุกแง่มุม

ภาพแสดงนักวิทยาศาสตร์ หลุยส์ ปาสเตอร์ และอีมิล คริสเตียน แฮนเซน (ภาพ AI สร้างโดย ผศ. ดร.ป๋วย อุ่นใจ)
เมื่อมนุษย์จะเขียนจีโนมยีสต์เสียใหม่
ในปี 2006 วิศวกรชีวภาพ เจฟ โบเก (Jeff Boeke) จากมหาวิทยาลัยจอห์นส์ฮอปกินส์ (Johns Hopkins University) เสนอไอเดียว่าอยากจะลองหาวิธีเขียนจีโนมของยีสต์ขึ้นมาใหม่ตั้งแต่ต้น เพราะยีสต์อยู่คู่กันมากับเผ่าพันธุ์มนุษย์มานับหมื่นปี เราใช้งานยีสต์มาอย่างสมบุกสมบัน แต่ความรู้จริง ๆ เกี่ยวกับจีโนมยีสต์นั้น เรายังไม่เข้าใจกันอย่างถ่องแท้ เจฟมองต่างมุมกับเครก เวนเทอร์และจอห์น กลาส เขาไม่ได้อยากได้ผ้าใบผืนว่าง จะได้แต่งเติมทุกอย่างจากศูนย์ เขาไม่ต้องการสิ่งมีชีวิตมินิมัล แต่เขาต้องการเขียนจีโนมของสิ่งมีชีวิตขึ้นมาใหม่จากศูนย์โดยใช้ยีสต์เป็นต้นแบบ เขาต้องการสร้างยีสต์เวอร์ชั่นใหม่ที่เขาเข้าใจหลักการการออกแบบ (design principle) ทุกอย่าง เป็นยีสต์เวอร์ชั่น 2.0 ที่หากเขาอยากดัดแปลงหรือแต่งเติมฟังก์ชั่นอะไรสักอย่างเข้าไป เขาจะสามารถทำได้เป๊ะตามที่เขาปรารถนา แม่นยำ รวดเร็วและมีประสิทธิภาพ ถ้าเปรียบ ทีม JCVI อยากได้ภาพสเก็ตที่มินิมัล ในขณะที่เจฟและทีม Sc2.0 ต้องการเข้าใจภาพทั้งภาพเพื่อที่จะได้เลือกเติม เลือกแต่งได้อย่างสัมฤทธิ์ผลสมประสงค์ แต่การจะเริ่มออกแบบและเขียนจีโนมของ “ยีสต์” ที่เป็นเซลล์ยูคาริโอตที่มีโครงสร้างที่ใหญ่ ยากและซับซ้อนขึ้นมาใหม่ตั้งแต่ต้นนั้นถือเป็นเรื่องที่มีแต่ในความฝัน ถ้าจะเป็นไปได้ ก็ต้องเป็นโครงการเมกะโปรเจ็คต์ขนาดใหญ่โตมโหฬารที่ต้องใช้ทุนรอนมากมายมหาศาล ไม่แพ้โครงการในตำนานอย่างโครงการถอดรหัสพันธุกรรมมนุษย์ (human genome project)
ก้าวแรกของเจฟ ถือเป็นความท้าทายยิ่งใหญ่ที่เขาจะต้องก้าวข้ามไปให้ได้ เพราะแค่เริ่ม รายจ่ายก็เบ่งบาน นักวิจัยเชี่ยวชาญที่จะมาช่วยก็ไม่มี ต้องยอมรับว่าแผนการที่เจฟวางไว้ช่างแยบยล แทนที่จะเริ่มด้วยการหาทางจ้างนักวิจัย (ที่จะทำงานได้มั้ย ไม่รู้) เจฟเลือกที่จะสร้างเเพลตฟอร์ม แล้วเปิดคลาสเรียนสร้างจีโนมขึ้นมาที่จอห์นส์ ฮอปกินส์ ในปี 2007 คลาสนี้มีชื่อว่า Build-a-Genome หรือ B-A-G คลาสเรียนนี้เริ่มต้นโดยมีหลักคิดว่าถ้าเราจะออกแบบจีโนมยีสต์เสียใหม่ “จะทำให้มันเรียบง่ายและควบคุมได้ดีกว่านี้ได้อย่างไร” การจัดการเรียนการสอนของคลาส B-A-G เป็นมากกว่าแค่สอนและให้ทำแบบฝึกหัดหรือแบบทดสอบในห้องเรียน แต่คือการสร้างแนวคิดสำหรับวิศวกรชีวภาพรุ่นใหม่

ภาพจำลองห้องเรียน B-A-G (ภาพ AI สร้างโดย ผศ. ดร.ป๋วย อุ่นใจ)
เพื่อให้ Sc2.0 ดำเนินไปอย่างเป็นระบบ เจฟได้นำเอาหลักการทางวิศวกรรม Design – Build – Test- Learn มาประยุกต์ใช้ในชั้นเรียนอย่างเคร่งครัด โดยเขาจะสอนให้นักศึกษาของเขาเริ่ม ออกแบบ (Design) ซึ่งจะเริ่มจากการวิเคราะห์ยีน ลบลำดับที่ไม่จำเป็น และเพิ่มจุดตัดต่อเพื่อการปรับแต่งในอนาคต และพอได้ลำดับดีเอ็นเอใหม่แล้ว พวกเขาก็จะ สร้าง (Build) ลำดับ DNA ที่ออกแบบนั้นขึ้นมาโดยการสังเคราะห์ทางเคมี และนำมาต่อเข้าด้วยกันเป็นสายยาว ต่อมา พวกเขาก็จะทดสอบ (Test) จีโนมใหม่นั้นในยีสต์ และประเมินว่าจีโนมที่ออกแบบมานั้นสามารถทำงานได้จริงในยีสต์หรือไม่ ท้ายที่สุด พวกเขาก็จะวิเคราะห์สรุปและประมวลผลการทดลองทั้งหมดที่ได้มาและเรียนรู้ (Learn) เพื่อนำไปปรับใช้ในการออกแบบลำดับดีเอ็นเอท่อนต่อ ๆ ไป ซึ่งรูบริกหรือเกณฑ์ประเมินก็ถูกปรับให้สะท้อนการคิดเชิงออกแบบ ความแม่นยำทางชีวสารสนเทศ และความร่วมมือในทีม ไม่ใช่แค่คะแนนสอบข้อเขียน
การจัดประสบการณ์การเรียนรู้ของเจฟน่าสนใจ การที่เขาเปิดโอกาสให้นักศึกษาร่วมเป็นผู้ออกแบบและสร้างโครโมโซมยีสต์เวอร์ชั่นใหม่ขึ้นมาจริง ๆ ได้ช่วยส่งเสริม ทักษะการคิดเชิงออกแบบและทำงานร่วมกันในทีมวิจัย อีกทั้งยังช่วยสร้างแรงบันดาลใจให้นักศึกษาหลายคนของเขาเกิดแรงฮึดและเติบโต จนเป็นแรงขับเคลื่อนสำคัญของวงการชีววิทยาสังเคราะห์ในปัจจุบัน
การจัดคอร์ส B-A-G ของเจฟกลายเป็นที่โจษขาน สำหรับกลุ่มลูกศิษย์และเพื่อนรวมงานของเจฟ Sc2.0 เป็นมากกว่าแค่คอร์สเรียนหรือโครงการวิจัย แต่มันเป็นฝันที่พวกเขามีร่วมกัน หลายปีผ่านไป คอร์ส B-A-G เริ่มกระจายไปในหลายมหาวิทยาลัย ในหลายประเทศมีนักเรียน นักศึกษาที่เคยผ่านคอร์สนี้มานับร้อย และท้ายที่สุด พวกเขาก็เริ่มก่อตั้งเป็น “ประชาคมวิจัย (research community)” เพื่อการเขียนจีโนมขึ้นมาใหม่ แต่โครงการนี้ก็ใช่จะราบรื่น มีหลายครั้งที่ การทดลองพวกเขาต้องหยุดชะงักด้วยปัจจัยหลายประการ เช่นการย้ายมหาวิทยาลัยของเจฟในปี 2013 และมีบางช่วงที่พวกเขาขาดเงินทุนวิจัย แต่ไม่ว่าจะเกิดอะไรขึ้น ก็ยังมีนักวิจัยแกนนำในประชาคมที่ยังพร้อมที่จะช่วยสนับสนุนและขับเคลื่อนโครงการนี้ต่อ
15 ปีกับความร่วมมือของประชาคมวิจัยทั่วโลกหลายร้อยชีวิต ในที่สุด ในปี 2025 จีโนมยีสต์ที่ถูกเขียนใหม่โดยมนุษย์ทั้งชุดก็เสร็จสมบูรณ์ เมื่อทีมวิจัยนำโดยมหาวิทยาลัยแมคแควรี (Macquarie University) ในประเทศออสเตรเลียได้รายงานความสำเร็จของการสังเคราะห์โครโมโซมสุดท้าย synXVI ในวารสาร Nature Communications และได้กลายเป็นอีกหนึ่งหมุดหมายที่สำคัญในระลอกคลื่นแห่งการพัฒนาเทคโนโลยีชีววิทยาสังเคราะห์ของมวลมนุษยชาติ ที่จะเปลี่ยนโฉมหน้าของชีววิทยาไปแบบกู่ไม่กลับ
นี่คือบทพิสูจน์ที่ชี้ชัดแล้วว่าถ้าเรามีศรัทธาและความรู้ “จีโนมของชีวิต” ก็ถูกลิขิตได้ด้วยมือน้ำมือมนุษย์ คำถามที่ต้องตอบให้ได้ เวลานี้ ก็คือเมื่อมนุษย์อหังการ์ถึงขนาดเขียนจีโนมของสิ่งมีชีวิตขึ้นมาได้แล้ว ในอนาคตข้างหน้า สังคมมนุษย์จะหาวิธีจัดการกับความเปลี่ยนแปลงและความเสี่ยงจากเทคโนโลยีพลิกโลกนี้ได้อย่างไร และเราจะเอาเทคโนโลยีสุดอลังการเหล่านี้มาใช้แก้ปัญหาอะไรกันบ้างเพื่อความกินดีอยู่ดีของมวลมนุษยชาติ
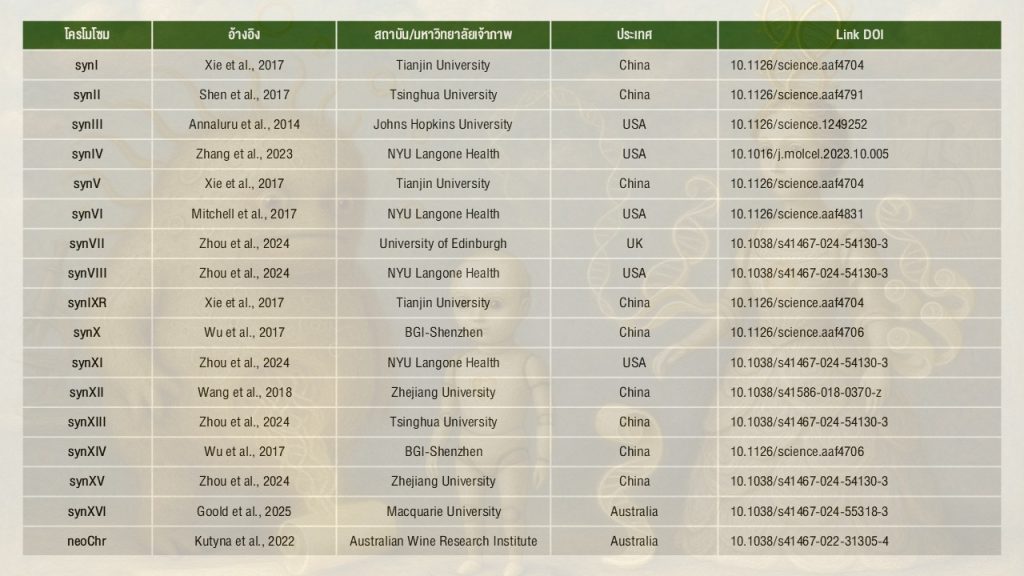
ตารางแสดงรายละเอียดของการมีส่วนร่วมในการเขียนโครโมโซมยีสต์ Sc2.0

References
ประวัติศาสตร์การใช้ยีสต์และการค้นพบในยุคก่อนชีวโมเลกุล
Schwann, T. (1837). Preliminary communication on the yeast plant and fermentation. Archiv für Anatomie, Physiologie und wissenschaftliche Medicin, 1837, 1–16.
Pasteur, L. (1876). Études sur la bière. Paris: Gauthier-Villars.
Synthetic Biology & Syn3.0 / Syn3A
Sc2.0: Synthetic Yeast Genome Project